ドラッグリパーパシング・サービス
Excelra独自の分析技術でドラッグリパーパシング(ドラッグリポジショニング)を加速
ドラッグリパーパシングの重要性はいくら強調してもし過ぎることはありません。
ドラッグリパーパシングまたはリポジショニングとは、既存薬に新たな治療機会を見出すことを目的とした医薬品開発戦略で、製薬企業やバイオテクノロジー企業にとって効果的かつ効率的であることが証明されています。従来のde novo創薬と比較すると、ドラッグリパーパシングは薬剤の上市に必要な時間とコストを大幅に削減します。
従来、ドラッグリパーパシングには2つの一般的なアプローチがありました。
1つ目は、薬物やターゲットを分析して代替用途を特定する「薬物またはターゲット中心のリパーパシング」です。
2つ目は、疾患のメカニズムを考慮し、そのメカニズムに影響を与える可能性のある既存の薬剤を特定する「疾患中心のリパーパシング」です。
近年では、薬剤の新たな組み合わせを見つけ、応用の幅を広げる第3のアプローチが勢いを増しています。
ずれのアプローチを取るにせよ、精密に選択され、効果的に分析されたデータの使用が、臨床試験へ進むドラッグリパーパシングの数を急速に増加させるベースとなっています。

医薬品中心的
医薬品をインプットとした代替疾患の特定に注力

ターゲット中心的
ターゲットをインプットとした代替疾患の特定に注力

疾患中心的
疾患をインプットとした代替薬/ターゲットの特定に注力
機械学習とデータ駆動型分析
高度な機械学習とデータ駆動型分析は、ドラッグリパーパシングに多大な影響を及ぼしています。生物学的実験によって生成されるデータの幅と深さが飛躍的に増大し、最新の分析ツールを用いることで、多様な方法でデータを照会し、解釈できるようになりました。
ドラッグリパーパシングにおける機械学習の利用
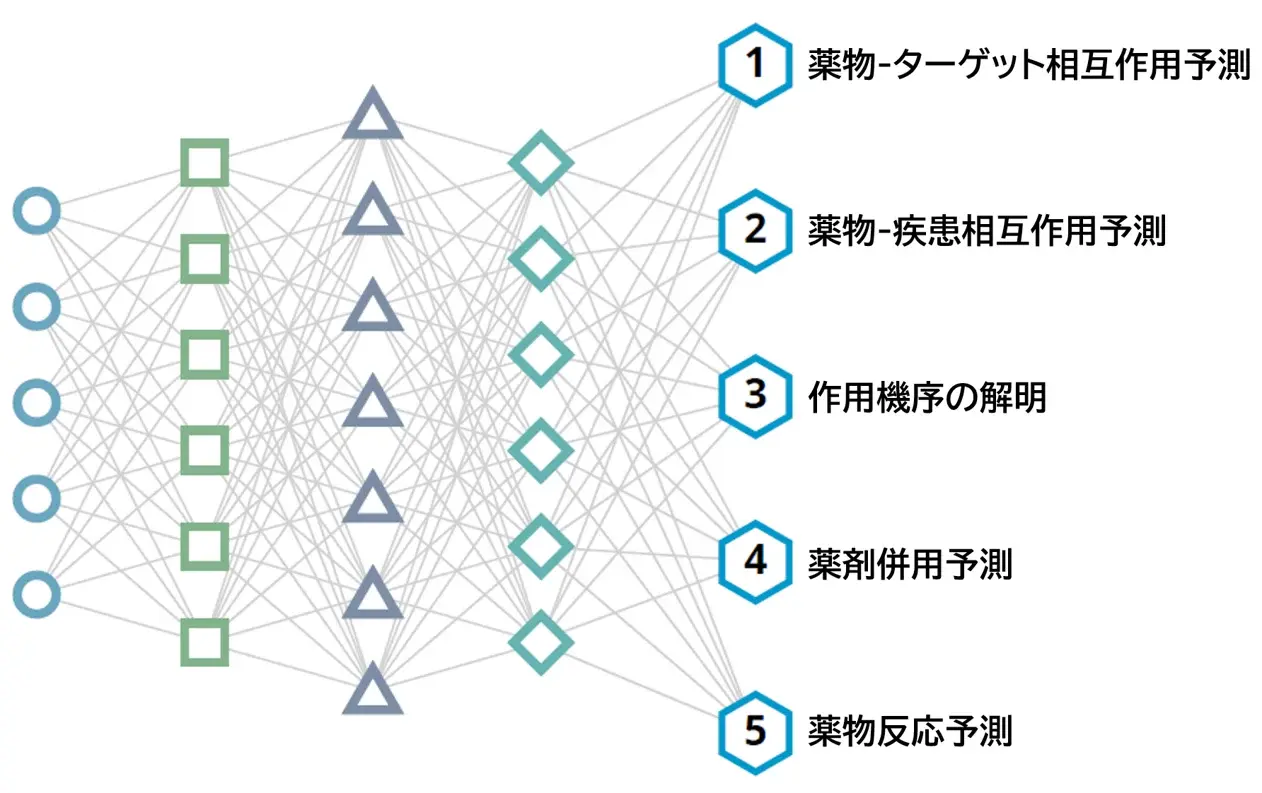
薬物-標的および薬物-疾患相互作用予測
ドラッグリパーパシングプログラムの基礎は、薬物、ターゲット、疾患間の関係を確立することにあります。OMICデータや生化学的アッセイエンドポイントなど、幅広いデータを活用することで、計算生物学者は薬物-標的および薬物-疾患の関連を同定し、正確に予測するための革新的な方法論を開発しました。これらの手法は、実験的にマップされた薬物ターゲットおよび薬物と疾患の相互作用ネットワークを補完し、ドラッグリパーパシングプログラムにおける貴重な追加要素となっています1。
1. 創薬標的相互作用予測
DTiGEMS+(Drug-target interactions using graph embedding, graph mining, and similarity-based techniques)は、異種ネットワークを薬剤予測グラフの形で構築する計算手法です。DTiGEMS+は、既知の薬物-標的相互作用のネットワークマップに、薬物-薬物の類似性および標的-標的の類似性を組み込んでいます。類似のアプローチとして、DGraphDTAがあります。DGraphDTAは、蛋白質と薬物の構造情報をマッピングするニューラルネットワークを使用して活性を予測します3。
2. 薬物-疾患相互作用予測
類似ネットワーク融合(SNF)とニューラルネットワーク(NN)の深層学習モデルは、薬物類似性、疾患類似性、既知の薬物-疾患相互作用のデータを活用して、薬物-疾患関連を正確に特定します。密畳み込みニューラルネットワーク(DCNN)は、薬物の構造的類似性と疾患の意味的類似性に関するデータを組み合わせた深層学習モデルのもう一つの例です。これらのモデルは、薬物と疾患の主要な特徴を捉え、それらの特徴の適応的最適化を適用することで、薬物と疾患の関連性をより適切に予測することができます5。
3. 薬物の作用メカニズムの解明
製薬会社の研究開発チームの主なタスクの一つは、薬剤や疾患の遺伝子シグネチャーを利用して、その作用機序(MoA)を評価することです。ネットワーク異常による作用機序の検出(DeMAND)は、MoAを評価するためのネットワークベースのアプローチの一例です。
また、遺伝子発現データを利用してディープラーニングにより作用機序をモデル化・予測するGPAR(Genetic Profile-Activity Relationship)ツールも存在します7。
4. 薬剤の組み合わせ予測
治療の有効性を向上させ、毒性を軽減するための新しい薬剤の組み合わせを適用することは、薬剤開発者にとって重要な目標です。しかし、適切な薬剤の組み合わせを特定するための実験的スクリーニングには多大な費用と時間がかかります。幸いなことに、SynGeNetのようなネットワークベースの計算アプローチや、DeepSynergyのような機械学習ベースのアプローチは、予測プロセスを劇的に加速させています。
9-11。
5. 薬物-疾患相互作用予測
腫瘍学における患者選択と層別化のための薬剤反応バイオマーカーの同定は、臨床試験の成功に直接影響するため、薬剤開発者にとって重要な関心事です。薬剤反応バイオマーカーを転帰と関連付けるには、十分なサンプルサイズ、効果的な計算ツール、およびコンテキストの認識が必要です。データが不十分な場合、機械学習の選択肢は限られていましたが、これらの障害を克服するために、治療に対する反応者と非反応者を分類するためのコンテキスト駆動型の単変量および多変量解析手法やディープラーニング手法がいくつか発表されています8。
高度なデータ解析と機械学習の応用には、細胞や組織を基にした反応予測やヒットの同定も含まれます。この分野は急速に発展しており、精度の向上、時間の短縮、そして従来の方法では見つけられなかった新規の関連性を明らかにする能力が期待されています。
ドラッグリパーパシング加速におけるExcelraの役割
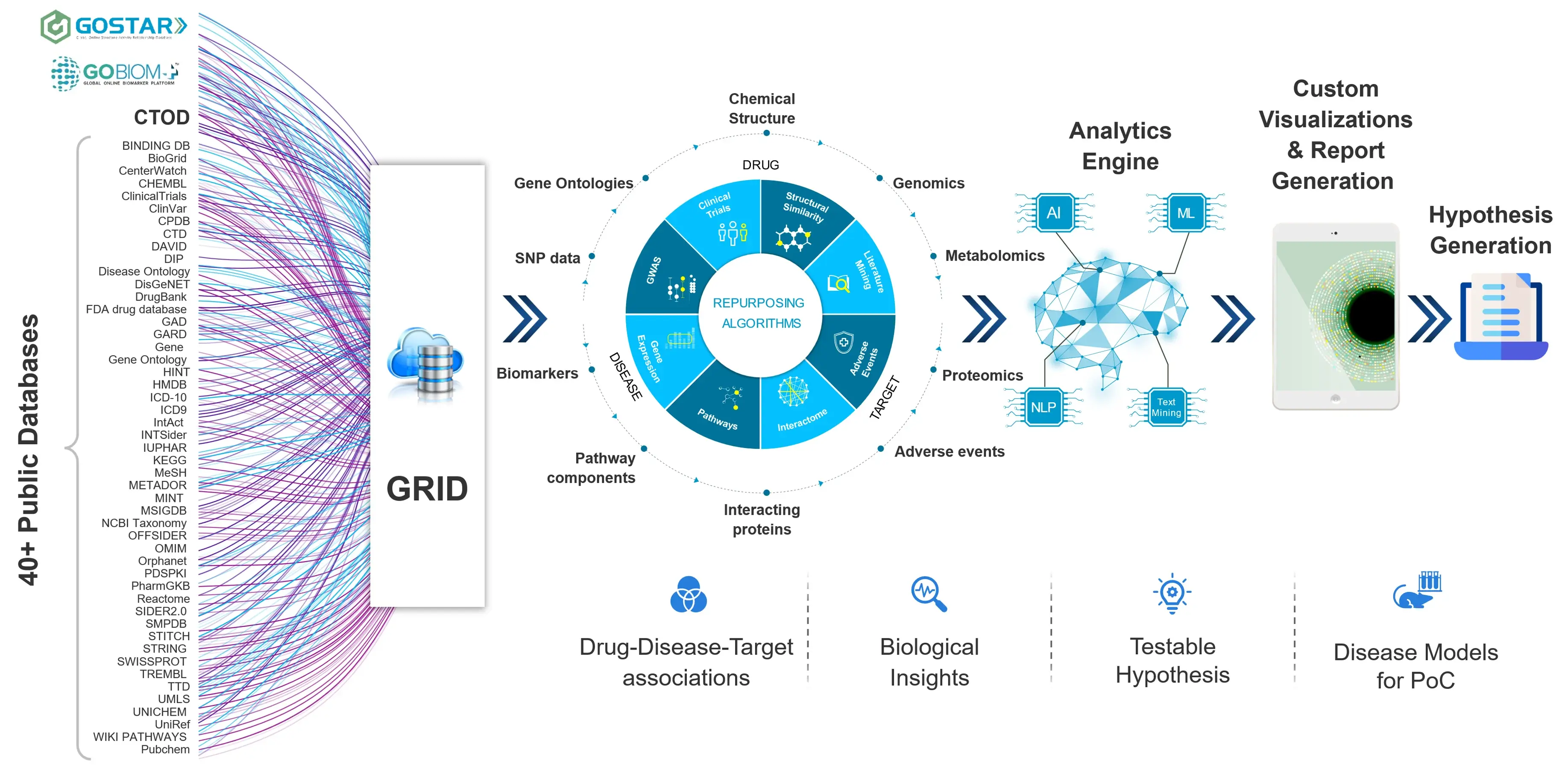
Excelraは、カスタムビルドのGlobal Repurposing Integrated Platform (GRIP) アルゴリズムを活用し、公共および独自のデータベースをマイニングして、高度な分析や予測機械学習モデル構築に使用するための高品質なデータを収集します。当社の専門家は、科学と技術の交差点で活動し、この2つの分野のギャップを埋めることに優れています。
Excelraは、データ革命の最前線に立ち、ドラッグリパーパシングプログラムに先進的なデータ分析技術を導入することを強く支持しています。データマイニング、データの構造化と変換、予測モデルの構築は、当社の専門技術の中核をなすものです。
Excelraは、専門的な知識と革新的な技術ソリューションを組み合わせ、生物学的な深い洞察と強力なデータ出力を提供します。これにより、クライアントにとって価値ある提案を行い、刺激的な成果をもたらします。
Excelraのドラッグリパーパシング・ソリューションは、柔軟でスケーラブルな分析パイプラインと、社内の専門家による手動データ駆動型分析を相乗的に組み合わせています。医薬品を化学的、生物学的、臨床的観点から総合的に分析し、新たな知見を見出します。
本サービスの分析プラットフォームGRIP(Global Repurposing Integrated Platform)は、Excelraの独自のSARデータベースGOSTAR(低分子構造活性相関データベース)、GOBIOM(バイオマーカー・データベース)、CTOD(臨床試験結果データ)に加え、40の公共データベースを統合し、自然言語処理や機械学習を用いてドラッグリパーパシングの為の分析を行います。GRIPには低分子医薬化合物、バイオマーカー、臨床試験データが含まれています。
実績・成功事例
これまでに積み重ねた成果が、私たちの信頼の証です。
年以上の低分子・バイオロジクス創薬の豊富な経験
Excelra独自のドラッグリパーパシング・プラットフォームGRIPを用いた包括的な分析
9
前臨床試験でバリデートされた開発候補
12
有望な結果を有する前臨床試験段階の開発候補
3
IND申請及び臨床試験に進んだ開発候補
ケーススタディー #1
目的
臨床化合物の代替適応を特定する
課題
米国西海岸のある製薬会社は、臨床候補化合物を再利用し、代替適応症を特定することを目指していました。
解決策
同社は、ExcelraのGRIPプラットフォームによって抽出されたデータを使用して、臨床化合物の代替適応症を特定しました。また、高度な機械学習モデルを活用してデータを処理し、医薬品と疾患の関連性を予測しました(図1参照)。
ExcelraのGRIPプラットフォームは、文献マイニング、臨床試験、構造類似性、インタラクトームによるインシリコ解析を組み込んでおり、薬物-薬物の類似性、疾患-疾患の類似性、標的作用機序に関連するパスウェイの特徴などを評価します。
潜在的な薬物-疾患ペアを同定するために、様々な機械学習アプローチが採用されました。これにより、一般に報告されているもの以外にも、関連性のある薬物-疾患間の関係を予測し、専門家の精査に役立てました。
結果
科学的根拠に裏付けられた5つの薬物-疾患関連の推奨が得られ、その中には2つの希少疾患、自己免疫疾患、消化器疾患、心血管疾患が含まれています。
創出された価値
Excelraのキュレーションされたデータは、代替の薬物-疾患ペアを評価するための高度なMLモデルで使用されました。このデータの鋭い応用により、多様な治療領域から顧客の臨床化合物の新規適応が導かれ、プログラムに大きな付加価値が生まれました。
-

図1.:薬剤-疾患または代替適応症の関連を特定するための機械学習へのアプローチ
サマリー
ドラッグリパーパシングは、入念に管理されたデータの利用可能性と、高度な分析および機械学習モデルの応用により、革命的な変革を遂げました。データを最大限に活用することで、新たな関連性の同定が進み、正確な解釈が飛躍的に加速しました。
参考文献
- Tanoli, , Vähä-Koskela, M., & Aittokallio, T. (2021). Artificial intelligence, machine learning, and drug repurposing in cancer. Expert opinion on drug discovery, 16(9), 977–989. https://doi.org/10.1080/17460441.2021.1883585
- Thafar, A., Olayan, R. S., Ashoor, H., Albaradei, S., Bajic, V. B., Gao, X., Gojobori, T., & Essack, M. (2020). DTiGEMS+: drug-target interaction prediction using graph embedding, graph mining, and similarity-based techniques. Journal of cheminformatics, 12(1), 44. https://doi.org/10.1186/s13321-020-00447-2
- Jiang, , Li, Z., Zhang, S., Wang, S., Wang, X., Yuan, Q., & Wei, Z. (2020). Drug-target affinity prediction using graph neural network and contact maps. RSC advances, 10(35), 20701–20712. https://doi.org/10.1039/d0ra02297g
- Jarada, N., Rokne, J. G., & Alhajj, R. (2021). SNF-NN: computational method to predict drug-disease interactions using similarity network fusion and neural networks. BMC bioinformatics, 22(1), 28. https://doi.org/10.1186/s12859-020-03950-3
- Wang, , Zhao, S., Zhao, J., & Feng, Z. (2021). A model for predicting drug-disease associations based on dense convolutional attention network. Mathematical biosciences and engineering : MBE, 18(6), 7419–7439. https://doi.org/10.3934/mbe.2021367
- Woo, J. H., Shimoni, Y., Yang, W. S., Subramaniam, P., Iyer, A., Nicoletti, P., Rodríguez Martínez, M., López, G., Mattioli, M., Realubit, R., Karan, C., Stockwell, R., Bansal, M., & Califano, A. (2015). Elucidating Compound Mechanism of Action by Network Perturbation Analysis. Cell, 162(2), 441–451. https://doi.org/10.1016/j.cell.2015.05.056
- Gao, , Han, L., Luo, D., Liu, G., Xiao, Z., Shan, G., Zhang, Y., & Zhou, W. (2021). Modeling drug mechanism of action with large scale gene-expression profiles using GPAR, an artificial intelligence platform. BMC bioinformatics, 22(1), 17. https://doi.org/10.1186/s12859-020-03915-6
- Adam, , Rampášek, L., Safikhani, Z., Smirnov, P., Haibe-Kains, B., & Goldenberg, A. (2020). Machine learning approaches to drug response prediction: challenges and recent progress. NPJ precision oncology, 4, 19. https://doi.org/10.1038/s41698-020-0122-1
- Güvenç Paltun, , Kaski, S., & Mamitsuka, H. (2021). Machine learning approaches for drug combination therapies. Briefings in bioinformatics, 22(6), bbab293. https://doi.org/10.1093/bib/bbab293
- Bai, L. Y., Dai, H., Xu, Q., Junaid, M., Peng, S. L., Zhu, X., Xiong, Y., & Wei, D. Q. (2018). Prediction of Effective Drug Combinations by an Improved Naïve Bayesian International journal of molecular sciences, 19(2), 467. https://doi.org/10.3390/ijms19020467
- Wang, , Li, H., & Guan, Y. (2020). Machine Learning for Cancer Drug Combination. Clinical pharmacology and therapeutics, 107(4), 749–752. https://doi.org/10.1002/cpt.1773
ケーススタディー #2
6種類の開発中止された化合物に対して30の新しい適応症を特定(大手製薬メーカー)
本ケーススタディでは、大手製薬企業がExcelraの支援を受け、開発が中止された6つの化合物に対して30の新しい適応症を発見したプロセスと結果を詳述しています。専用リポジショニングプラットフォーム「GRIP」とオミクス分析を駆使し、これらの化合物の新たな可能性を見出し、実験で検証しました。ご興味のある方は、ぜひダウンロードして詳細をご覧ください。新たな医薬品開発の一助となる貴重な情報が満載です。